シーケンサーから出力されるリードデータにはアダプター配列、ポリ A、ポリ T、低クオリティリードなどが含まれている場合がある。そのため、解析を行う前に、データのクオリティなどを一度チェックして、必要に応じてクリーニングしていく必要がある。このような作業をクオリティコントロールなどと呼んだりする。
FASTQ のクオリティコントロールでは、アダプター配列やポリ A、ポリ T と思われる部分を除去し、低クオリティのリードをフィルタリングしていく。クリーニング後の FASTQ のクオリティをもう一度チェックし、必要に応じて再クリーニングする。FASTQ のクオリティが十分に信頼できるようになるまでこれを続ける。
このようにクオリティのチェックやクリーニングには様々なプログラムが用意されている。よく知られているものとしては FastQC や FASTX toolkit などがある。
| プログラム | 言語 | クオリティチェック | アダプター除去 | ポリ A/T テール除去 | トリミング | フィルタリング |
| FastQC | Java | ✓ | ||||
| cutadapt | Python | ✓ | ||||
| Trimmomatic | Java | ✓ | ✓ | |||
| PRINSEQ | Perl | ✓ | ✓ | ✓ | ✓ | |
| FASTX-toolkit | C | ✓ | ✓ | ✓ | ✓ |
リードクオリティに関する疑問など
クオリティスコアと信頼度
クオリティスコアは Phred 形式が利用されている。シーケンシングエラーが生じる確率 perror に対し、クオリティスコアは -10log10perror によって計算される。スコアからは以下のことが言える。(詳細:クオリティスコアについて)
- クオリティスコアが 10 ならば、シーケンシングエラーが生じる確率は 10.0% であるから、読み取られた塩基の信頼度は 90.0% である。
- クオリティスコアが 20 ならば、シーケンシングエラーが生じる確率は 1.0% であるから、読み取られた塩基の信頼度は 99.0% である。
- クオリティスコアが 30 ならば、シーケンシングエラーが生じる確率は 0.1% であるから、読み取られた塩基の信頼度は 99.9% である。
3' 末端のクオリティスコアが低い理由
シーケンシング反応は 5'→3' 方向に、次のように行われる。
- 保護基の付いた dNTP(dATP、dGTP、dCTP、dTTP)のうち 1 種類だけ反応系に加える
- DNA ポリメラーゼにより伸長反応を行う(dNTP には保護基が付いているため dNTP が取り込まれると伸長反応が停止する)
- 未反応の dNTP を除去する
- 蛍光標識を検知する
- 保護基と蛍光標識を取り除く
基本的には dNTP を変えながらこのサイクルを繰り返すことによって伸長反応を行う。こうした反応では、dNTP がきれい除去できなかったり、保護基がうまく取り外せなかったりする、などのエラーが生じる可能性がある。このようなエラーはサイクルを繰り返すたびに積み重なる。これが 5' 末端から離れば離れるほど、リードのクオリティスコアが低下する原因となる。
(参照:SEQanswers, illumina 1塩基合成反応)
クオリティスコアの閾値の決め方
クオリティスコアを利用したリードのクリーニングの際に、閾値を目的に応じて変更する必要がある。発現量の比較検定が目的の場合は低めの閾値にしても構わないが、SNP 検出などが目的の場合は高めの閾値にする必要がある。また、比較的最近のシーケンサー(HiSeq 2000 など)ならば、クオリティが 30 を満たさないものを低クオリティとして取り扱う場合が多い(一昔のものでは 20 を閾値とする場合が多いかった)。
5' 末端の 13 塩基の各位置における出現頻度について
RNA-seq を利用して遺伝子の発現量を定量するとき、mRNA など細胞から抽出し、断片化する必要がある。断片化が mRNA 上の任意の位置でランダムに行われるのであれば、断片上のすべての位置において塩基 A の出現確率が等しいと考えられる。同様に C、G、T についても同様に考えられる。しかし、シーケンサーを利用して読み込んだ断片では、最初の 13 塩基が明らかにこのような傾向がみられない。例えば、以下のグラフは横軸が断片の位置、縦軸が各塩基の出現確率を表している。1-13 塩基と 13 塩基以降では明らかに傾向が異なる。(T:赤、C:青、A:緑、G:黒)
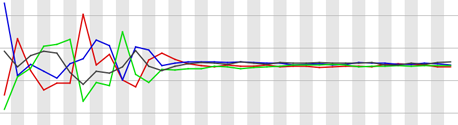
このパターンは再現可能で、異なる研究機関や研究室で RNA-seq 解析を行っても同様なパターンが観測される。一方、DNA-Seq などでは観測されていない。このパターンが観測される原因は DNase I にあると考えられる。昔に試料調製を行うときは、mRNA 抽出、cDNA 合成、cDNA の断片化の順で行っていた。cDNA の断片化の際に DNase I を利用した場合、このようなパターンが観測される。 一方、音波破砕などにより CDNA を断片化するとき、このパターンが見られない。そのため、このパターンを取り除くべきか、取り除くべきではないかで意見が分かれるところである。
(参照:Kasper et al.)